Wissenschaftliches Konzept
(Anm.: Unter „Mikrobiom“ versteht man die Lebensgemeinschaft aller Mikroorganismen, die ein Lebewesen besiedeln)
1.) Forschungsziele
Mikrobiome sind stark mit unserer Gesundheit assoziiert; das Wissen darum ist allerdings noch stark limitiert.
Kürzlich entwickelte Hochdurchsatzsequenzierungs-Techniken ermöglichen uns, bislang unerforschte Habitate bezüglich ihrer Mikroorganismen zu erforschen.
Wir haben hierzu Äpfel ausgewählt, da ihr Mikrobiom bislang international nicht untersucht wurde, aber einen starken Einfluss auf die menschliche als auch auf die Pflanzengesundheit ausüben kann.
Im Rahmen des Projektes soll nach umweltfreundlichen Lösungen für Apfelkrankheiten und Lagerungsprobleme gesucht werden. Die Forschungsziele werden durch aktive Mitwirkung von SchülerInnen erreicht. Hier ergibt sich eine wichtige Möglichkeit, aktuelle Forschung vom international anerkannten Standard mit voruniversitärer Nachwuchsförderung zu verbinden. Die SchülerInnen lernen dabei nicht nur wissenschaftliche Vorgehensweisen, sondern ermöglichen durch den Zugang zu Äpfeln aus Privatgärten bzw. Lebensmittelgeschäften ein breiteres Hochdurchsatzscreening von Pflanzenkrankheitsantagonisten.
Die Zusammensetzung von Apfel-assoziierten Mikrobiomen, die aus ökologischem und herkömmlichem Anbau stammen, soll mit Hilfe von mikro- und molekularbiologischen bzw. mikroskopischen Methoden verglichen werden. Zusätzlich sollen Unterschiede in Apfelmikrobiomen von krankheitsbefallenen und gesunden Apfelbäumen sowie von Äpfeln die unterschiedlich gelagert wurden, zum ersten Mal erfasst werden.
Ein wichtiges Ziel des Projektes ist die Vermittlung der Wissenschaft und deren Umsetzung in unserer Gesellschaft. Dies wird nachhaltig durch begleitende Kunstprojekte erreicht, wodurch Forschungsthemen den SchülerInnen und der interessierten Bevölkerung näher gebracht werden. Dieser innovative interdisziplinäre Ansatz wäre ohne die Unterstützung des Sparkling Science Programms nicht durchführbar.
2.) Stand des Wissens
A. Zur Entdeckung von Mikrobiomen.
Während einzelne Mikroorganismen (MO) bereits nach der Erfindung des Mikroskops im 17. Jahrhundert durch Antoni van Leeuwenhoek sichtbar gemacht werden konnten, blieben die Identität der einzelnen Mikroorganismen sowie die Struktur komplexer mikrobieller Gemeinschaften lange Zeit unbekannt.
Erst Ende des 20. Jahrhunderts wurde die Technik entwickelt, die es erlaubt, molekulare Fingerprints von Mikroorganismen-Gemeinschaften abzubilden (Muyzer & Smalla 1998). Diese Technik rief bereits eine erste Blüte der mikrobiellen Ökologie hervor, da man nun damit mehr (ca. 20 %) der existierenden Mikroorganismen erfassen konnte. Mit bis dahin üblichen Kultivierungsmethoden waren maximal 3 % von ihnen nachweisbar.
Erst die Hochdurchsatzsequenzierung von DNA machte es möglich, das volle Ausmaß der mikrobiellen Vielfalt zu erfassen.
Studien, die in den letzten zehn Jahren durchgeführt wurden, ergaben eine hohe Anzahl von überraschender Vielfalt in den mikrobiellen Ökosystemen. Im menschlichen Darm leben zum Beispiel mehr als 100 Trillionen Mikroorganismen; eine Zahl, die zehnmal größer ist als der menschliche Körper Zellen besitzt (Qin et al. 2010). Die Artenzahl von Mikroorganismen, die den menschlichen Körper besiedeln, wird auf mehr als 10.000 geschätzt.
Ähnliche Dimensionen werden für Pflanzen berichtet: in der Pflanzenwurzel, die ähnlich wie unser Darm der Nährstoffaufnahme dient, leben mehr Mikroorganismen, als Menschen auf der Erde (Philippot et al. 2013).
Die komplexen Mikroorganismen-Gemeinschaften werden als Mikrobiome bezeichnet. Neben dieser beeindruckenden Vielfalt tragen die wichtigen Funktionen von Mikroorganismen zu einer neuen Sichtweise bei (Lozupone et al. 2012).
Der Imagewechsel der Mikroorganismen kann als Paradigmenwechsel bezeichnet werden: alle Eukaryoten definieren sich als Meta-Organismen und sollten nur im Zusammenhang mit ihrem Mikrobiom betrachtet werden (Jones 2013). Weiters zählt dazu auch die Entdeckung, dass Pathogene nur einen verschwindend geringen Teil der Mikroorganismen darstellen; oftmals ist hier nur die Balance verschoben. Wissenschaftler fordern deshalb ein Umdenken im Umgang mit Mikroorganismen (Blaser 2011). Dieses Umdenken ist auch für die Bevölkerung wichtig und sollte wissensbasiert erfolgen.
Erste Erkenntnisse legten eine bislang große und oft unerwartete Vielfalt in Mikrobiomen offen, allerdings sind die beeinflussenden Faktoren oft nicht bekannt. Insbesondere im Zusammenhang mit Lebensmitteln stellt die Anwendung von Mikroorganismen zum Pflanzenschutz und zur Stressabwehr derzeit einen Umbruch in der Landwirtschaft dar.
Klassische Monitorings im Lebensmittelbereich erlauben nur einen geringen Anteil der Diversität zu bewerten und liefern somit keine repräsentativen Aussagen. In der Erforschung gesunder Lebensmittel mit weniger Pestiziden liegt somit ein großes Potenzial für die Entwicklung von gesunder Nahrung.
Es ist für uns von besonderer Bedeutung diese Zusammenhänge zu erforschen und sie für die Jugendlichen sowie die breitere Bevölkerung verständlich und greifbar zu machen.
B. Der Apfel als Forschungsobjekt für unsere Gesundheit.
Bei vielen Krankheiten wie Herz-Kreislaufkrankheiten, Allergien und Krebs wurde eine Mitbeteiligung des menschlichen Mikrobioms festgestellt (Qin et al. 2010).
Auch die Pflanzengesundheit wird nun im Zusammenhang mit den assoziierten Mikroorganismen gebracht: ohne sie sind viele nicht in der Lage zu keimen, zu wachsen oder wohlschmeckende Früchte zu bilden (Berg et al. 2013, 2016).
Interessanterweise gibt es einen Zusammenhang zwischen den beiden Mikrobiomen: unsere Nahrung bestimmt nicht nur direkt über ihre Zusammensetzung unser eigenes, individuelles Mikrobiom, sondern dient auch als wichtige Quelle für nützliche Mikroorganismen (Thomas et al. 2011). Obwohl diese Tatsache wichtig für ein gesundes Darmmikrobiom ist und helfen kann, Krankheiten wie Entzündungen und Adipositas zu verhindern (Le Chatelier et al. 2013), sind die Mikrobiome auf unseren Lebensmitteln bis auf wenige Ausnahmen unbekannt (Leff und Fierer 2013).
In dem neusten Report von dem National Science and Technology Council (November 2015) wurde die Mikrobiom-Untersuchung von Lebensmitteln als Forschungsschwerpunkt Nummer eins in der internationalen Mikrobiomforschung bezeichnet. In westlichen Ländern stellen Äpfel neben Trauben und Orangen eine wichtige Quelle für die Aufnahme von Obst aus der Nahrung dar (Perez et al., 2001). Epidemiologische Studien deuten auf eine inverse Korrelation zwischen regelmäßigen Verzehr von Äpfeln und dem Risiko für unterschiedliche Krebserkrankungen, Herz-Kreislauf Erkrankungen, sowie Typ II Diabetes hin (Jedrychowski et al. 2009).
Die gesundheitsfördernden Eigenschaften von Äpfeln wie auch anderen pflanzlichen Lebensmitteln die roh verzehrt werden, könnten unter anderen auch an dem dazugehörigem Mikrobiom liegen. Die Mikrobiome der Apfelbaumrhizosphäre (unmittelbar durch eine lebende Wurzelbeeinflussten Raum im Boden) sowie von Apfelblüten wurden in den letzten Jahren durch einige Arbeitsgruppen gründlich erforscht (Shade et al. 2013; Caputo et al 2015). Uns ist aber keine Studie bekannt die sich mit den Mikrobiomen von Apfelfrüchten beschäftigt, was die Innovation und Relevanz unseres Vorhabens nochmals unterstreicht.
Eine der häufigsten Krankheiten von Apfelbäumen im europäischen Raum ist der Apfelschorf (MacHardy 1996; Abb. 1), der durch den Schimmelpilz Venturia inaequalis verursacht wird und zu Ertragsverlusten von 70 % und höher führen kann.
Gloeosporium-Fäulen am Apfel ist eine wichtige Lagerkrankheit, die von drei unterschiedlichen Pilzen hervorgerufen werden kann. In den vergangenen Jahren wuchs die Resistenz von Venturia gegenüber einer zunehmenden Anzahl von chemischen Fungiziden in großen Apfelanbaugebieten.
Eine umweltfreundliche Alternative zu chemischen Pestiziden ist die Biokontrolle, ein Prozess in dem man lebendige Organismen nutzt um Schädlinge zu bekämpfen (Berg 2009).
An der biologischen Kontrolle von Apfelschorf wurde seit 50 Jahren geforscht (Carisse 2000). In einer neuen Studie wurde gezeigt, dass ein Pilz (Cladosporium cladosporioides H39) in der Lage ist die Sporenbildung von V. inaequalis deutlich zu reduzieren. Es konnte sogar eine Wirkung von H39 in Feldversuchen in kommerziellen Obstgärten nachgewiesen werden (Köhl et al. 2015). Nichtsdestotrotz gibt es noch keine mikrobiologischen Produkte gegen diese Krankheit auf dem Market (Carisse 2000; Köhl et al. 2015).
3.) Wissenschaftliche
Herangehensweise
Wir wollen uns mit Hilfe von mikro- und molekularbiologischen bzw. mikroskopischen Analysen auf die Spurensuche nach der Vielfalt mikrobiologischer Gemeinschaften auf und in biologisch und konventionell gewachsenen, gesunden und krankheitsbefallenen Äpfeln begeben.
Viele der ausgewählten Methoden wurden in der Arbeitsgruppe von Univ. Prof. Gabriele Berg entwickelt, die im Bereich der pflanzlichen Mikrobiomforschung international anerkannte Expertise besitzt.
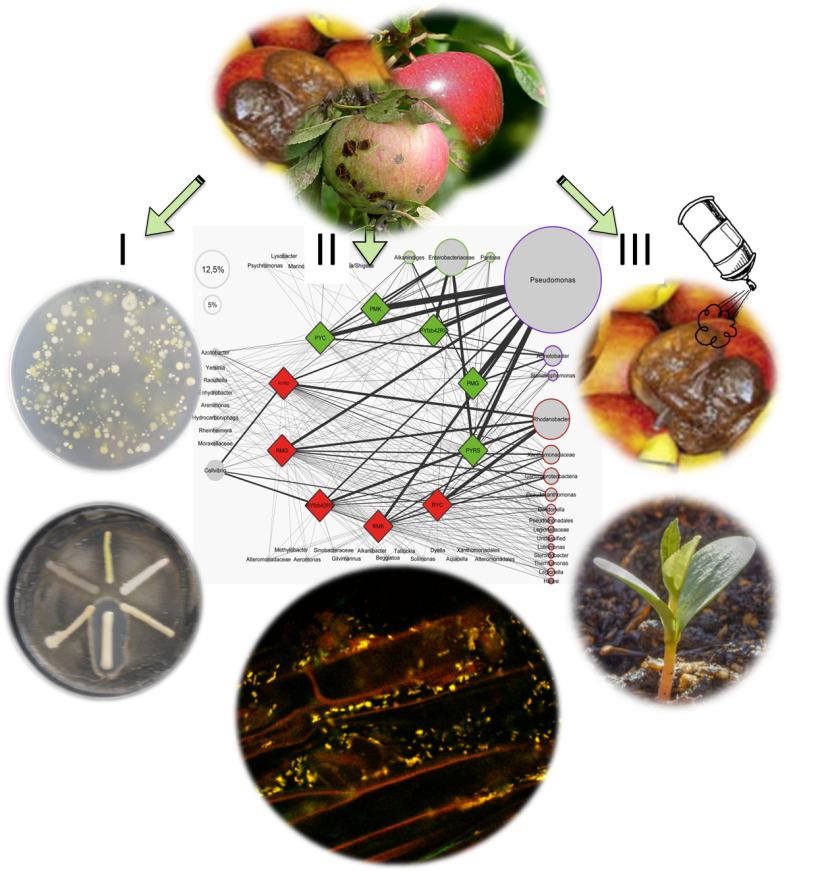
Die im Projekt geplanten Experimente sind in der Abb. 1 dargestellt. Die experimentellen Aufgaben werden mit ausgewählten SchülerInnen in Kleingruppen unter Betreuung durch MasterstudentInnen und InstitutsmitarbeiterInnen durchgeführt, wobei bei den Modulen 1 und 6 die Gesamtklassen zu dem Projektergebnissen beitragen werden (siehe „Methoden der Zusammenarbeit“).
Abb. 1.
Schema der
wissenschaftlichen Herangehensweise.
Die Mikroorganismen von gesunden Äpfeln, sowie von mit Apfelschorf bzw. Apfelfäule erkrankten Äpfeln werden mit drei unterschiedlichen Methoden untersucht:
(I)
Kultivierungs-abhängige Methoden beinhalten die Isolierung der einzelnen bakteriellen Stämme auf einer Agarplatte und den anschließenden Hemmhoftest um die Antagonisten der Pflanzenkrankheiten zu selektieren.
(II)
Durch die kultivierungs-unabhängigen Methoden wird die Gesamtheit der Mikroorganismen im Apfel erfasst. Die bakteriellen Netzwerke werden mithilfe bioinformatischer Methoden visualisiert (Abb. aus Erlacher et al. 2014). Die Konfokale Laserscanning-Mikroskopie erlaubt es, die Kolonisierungsmuster von natürlichen auf dem Apfel gewachsenen Mikroorganismen zu veranschaulichen.
(III)
In Pflanzenmodelltests werden die ausgewählten Bakterienstämme auf ihre Wirksamkeit gegen Pflanzenkrankheiten in planta getestet.
Anschließend werden umweltfreundliche antimikrobielle Substanzen gegen Lagerungskrankheiten getestet.
Modul 1
Ökologische Exkursionen (Jahre 1 und 2)
Bei den Besuchen in SEKEM‘s Obstgartenanlage wird das Wissen über die nachhaltige Agrarwirtschaft durch vielfältige hands-on Aktivitäten vermittelt und den SchülerInnen dadurch Probleme und Lösungen nahe gebracht. Es werden Pflanzen untersucht und Blattkrankheiten gezeigt, sowie Bodenprofile vorgestellt. Das gesammelte pflanzliche Material wird dazu genutzt, um kultivierbare Mikroorganismen, die in den Äpfeln leben, auf Agarplatten zu züchten sowie die Gesamtheit der Mikroorganismen durch Sequenzierungstechniken zu erfassen. Dieses Modul wird direkt mit dem Modul 6 verknüpft indem die SchülerInnen die Probenahmeorte detailliert dokumentieren, die ausgesuchte Pflanzenarten fotografieren und zeichnen und auf diesem Weg über sie lernen.
Modul 2
Neue Mikroorganismen für Pflanzenschutz (Jahr 1)
Die im Modul 1 aus Äpfeln isolierten Bakterien werden von ausgewählten SchülerInnen im Labor der TUG hinsichtlich ihrer Aktivität gegen den Apfelschorf Erreger Schimmelpilz V. inaequalis, sowie das Pflanzenpathogen Verticillium longisporum untersucht (Rybakova et al. 2015; Fiss at al. 2000). Alle in den Untersuchungen benutzten Stämme sind für die menschliche Gesundheit unbedenklich. Wirksame Isolate werden als Antagonisten bezeichnet und können unter anderem durch die Bildung eines Hemmhofes in einem Dualkulturtests identifiziert werden. Solche Mikroorganismen, die in der Lage sind V. inaequalis und/oder V. longisporum zu hemmen, werden ganz genau mikroskopisch und genetisch untersucht (DNA Sequenzierung).
Folgende Fragen können damit beantwortet werden:
1.) aus welchen Lebensmitteln bzw. Pflanzen wurden mehr Antagonisten gewonnen? und
2.) können Unterschiede in der Anzahl von Antagonisten bei Lebensmitteln aus biologischen und konventionellen Anbau nachgewiesen werden?
Modul 3 (Jahr 1)
Analyse des Apfelmikrobioms
Apfelbäume aus ökologischem Anbau (Firma SEKEM ENERGY GmbH) sowie konventionell gezogene Äpfeln und solche aus privaten Gärten der SchülerInnen werden auf Krankheitsbefall untersucht.
Es werden zusätzliche Parameter erfasst, wie z.B.:
1.) Herkunft und Produktionsweise: biologisch, konventionell, private Gärten oder Großanbau
2.) Alter
3.) Qualität und Pathogenbefall;
4.) Lagerung: frisch geerntet oder gelagert. Diese Parameter können dann mit den Daten statistisch verglichen werden, um den wichtigsten Einfluss auf die Mikrobiom-Zusammensetzung zu identifizieren.
Die Aufarbeitung der DNA Proben umfasst folgende Schritte
1.) DNA-Isolierung und
2.) Vervielfältigung mittels PCR mit spezifischen Primer für Bakterien und Archaea (515F/806R, Caporaso et al. 2012),
3.) Sequenzierung der DNA,
4.) bioinformatorische Auswertung.
Die wissenschaftlichen Methoden sind in Bragina et al. (2013) und Erlacher et al. (2014) ausführlich dargestellt. Die Mikroorganismen im Apfel werden zusätzlich mit Hilfe von konfokaler Laserscanning-Mikroskopie visualisiert. Hierzu werden die Proben zunächst fixiert und dann mit fluoreszierenden DNA-Sonden markiert. Außerdem kann durch die Anwendung von LIVE/DEAD® BacLight Kit’s (ThermoFisher) zwischen lebenden und toten Bakterien unterschieden werden und somit die Ergebnisse der Mikrobiomuntersuchung entsprechend ergänzen. Als Ergebnis der wissenschaftlichen Untersuchung stehen Bakterienbibliotheken, Netzwerkanalysen sowie ein Bildarchiv mit lasermikroskopischen Aufnahmen von Mikroorganismen der Äpfel zur Verfügung.
Die bioinformatischen Daten werden in internationalen Datenbanken abgelegt, wo sie von Forschern aus aller Welt verwendet werden können und für weitere Forschungsprojekte zur Verfügung stehen.
Mit den gewonnenen Daten werden wir unter anderem folgende Fragen beantworten: 1.) welche Mikroorganismengruppen leben in den Äpfeln, die wir essen? Wie viele davon leben zum Zeitpunkt des Konsums?
2.) Wie unterscheiden sich die Mikrobiome von Äpfeln aus biologischem und konventionellem Anbau, von Privatgärten und Großanbau, sowie von krankheitsbefallenen und gesunden Bäumen?
3.) Welche und wie viele dieser Mikroorganismen können als potenziell pathogen, oder nützlich für Mensch bzw. Pflanze angesehen werden und was bedeuten diese Unterschiede für unsere Gesundheit?
4.) Gibt es Indikatoren für gesunde Apfelbäume?
5.) Welche Mikroorganismen kommen am häufigsten in den kranken Apfelbäumen vor? Diese Mikroorganismen werden dann in den Folgeprojekten (Modul 5 und Sommerpraktikum) gezüchtet.
Modul 4 (Jahr 2)
Neue Mikroorganismen gegen Lagerkrankheiten
Es werden Mikroorganismen aus den von Fäule-Erregern befallenen Äpfeln isoliert. Zusammen mit den ausgewählten Bakterien vom Modul 1 und aus den Sommerpraktika werden sie auf ihre Fähigkeit geprüft, die Gloeosporium-Fäule Erreger Gloeosporium album and Gloeosporium perennans, auf einer Agar Platte zu hemmen.
Modul 5 (Jahr 2)
Testung von Mikroorganismen in Pflanzenmodellen
Die in den Modulen 1 und 4 isolierten Antagonisten von pflanzlichen Pathogenen werden zusätzlich in Pflanzenmodellen gegen folgende Krankheitserreger wie in der entsprechenden Literatur beschrieben getestet:
1.) gegen Apfelfäule-Erreger im Apfelmodell (Weiss et al 2006);
2.) gegen Venturia inaequalis im Apfelblatt-Modell (Yepes et al. 1993); und
3.) gegen Venturia inaequalis im Apfelkeimling-Modell (Fiss et all, 2000). Die Mikroorganismen werden zusätzlich mit Hilfe von konfokaler Laserscanning-Mikroskopie in vivo visualisiert.
Sommerpraktika / Maturaarbeiten
Bewertung potentieller Anwendungen gegen Pflanzen- und Lagerkrankheiten. Dieses Modul wird im Rahmen eines Sommerpraktikums von zwei SchülerInnen (Maturaarbeiten) durchgeführt und von Mitarbeitern der TUG sowie der Firma Roombiotic GmbH in Zusammenarbeit mit der Lagerungsfirma Meleco betreut.
Äpfel aus biologischer Herkunft, werden mit einem biologischen antimikrobiellen Mittel natürlicher Abstammung, das von der Firma Roombiotic erforscht wird, behandelt (Abb. 1).
Nach anschließender Lagerung unter kommerziellen Bedingungen werden die behandelten Äpfel auf das Vorhandensein von Lagerungskrankheiten untersucht und mit einer unbehandelten Kontrollgruppe verglichen.
Anschließend werden die befallenen Äpfel aussortiert und für die Isolation von Mikroorganismen die mit der Krankheit assoziiert sind, verwendet.
Die Mikroorganismen werden gezüchtet und deren 16S rRNA Genfragment sequenziert. Dabei werden die Ergebnisse der bioinformatischen Auswertung (bakterielle Netzwerke; Modul 2) benutzt, um die Bakterien auszuwählen, die mit der Krankheit assoziiert sind.
Diese ausgewählten Stämme werden gezüchtet und auf Sensitivität gegenüber dem biologischen antimikrobiellen Mittel getestet.
Dadurch wird ein breiteres Hochdurchsatzscreening nach nützlichen Mikroorganismen (Antagonisten von Pflanzenpathogen) ermöglicht was wiederum eine umfassende Qualitätskontrolle der gelagerten Äpfel gewährleistet.
Die gewonnenen Daten werden von der Firma Roombiotic verwertet sowie im Rahmen einer oder mehreren Maturaarbeit/en bearbeitet.
Modul 6 (Jahre 1 und 2)
Künstlerisch-mediale Umsetzung
Die SchülerInnen werden im Anschluss zu ihren Forschungsarbeiten innerhalb des Projektes an einem professionell betreuten EcoArt Kunstprojekt teilnehmen, das ihre Ergebnisse für die breitere Öffentlichkeit greifbar machen wird und auf besonders großes mediales Interesse stoßen soll.
Damit wird ein einzigartiger Wissenstransfer ermöglicht, der alle Altersstufen und Bevölkerungsschichten erreicht.
Die EcoArt Ausstellung wird zweimal, jeweils zum Semesterende stattfinden und vom professionellen Künstler, Bildhauer und Erzieher Timothy Mark organisiert und gestaltet.
Die Konfokalmikroskopiebilder, die im Rahmen des Projektes entstehen, werden vergrößert und kreativ als Kunstobjekte dargestellt. Zusätzlich wird eine dreidimensionale Installation mit Hilfe der SchülerInnen unter professioneller Einleitung und Beaufsichtigung von Herrn Mark angefertigt, die die Kolonisierung der Pflanze mit den Mikroorganismen darstellen soll.
Diese Skulptur wird an einem öffentlichen Platz (z.B. Botanischen Garten in Graz) aufgestellt, um zur Interessensanregung der Bevölkerung zu dienen. Dadurch kann Unsichtbares in eine begreifliche Form sichtbar gemacht werden. Darüber hinaus soll ein neuer Zugang zu wissenschaftlichen Erkenntnissen geschaffen werden, der dem jüngeren Publikum das spielerische Erforschen von umweltmikrobiologischen Fachgebieten ermöglicht und das Nutzen von Mikroorganismen für unsere Gesundheit erklärt.
Es ist geplant, dass Gesamtklassen, LehrerInnen, interessierte Eltern und Geschwister, sowie Freunde von den Jugendlichen an der Ausstellungsvorbereitung aktiv teilnehmen werden.
Der EcoArt Day wird vor allem dazu dienen, wissenschaftliche Ergebnisse kreativ darzustellen. Dabei lernen die SchülerInnen außer PowerPoint zu gestalten, auch andere Medien zu benutzen, wie selbst produzierte Bilder, 3D Objekte, Filme, Musik und Ähnliches zu benutzen.
Um den TeilnehmerInnen und Gästen Einblicke in den wissenschaftlichen Alltag zu ermöglichen und die vorgetragene Forschungsthemen besser zu verstehen, werden dem Publikum während des EcoArt Days spannende hands-on Experimente angeboten (Stationslauf).
Dies wird von der Firma Biotenzz zusammen mit der TUG veranstaltet.
Literaturliste
Berg G (2009) Plant–microbe interactions promoting plant growth and health: perspectives for controlled use of microorganisms in agriculture. Applied microbiology and biotechnology 84(1):11-18
Berg G, Zachow Z, Müller H, Philipps J, Tilcher R. (2013) Next-generation bio-products sowing the seeds of success for sustainable agriculture. Agronomy 3: 648-656
Berg G, Rybakova D, Grube M, Köberl, M. (2016) The plant microbiome explored: implications for experimental botany. J Exp. Botany 67:995-1002
Blaser M. 2011. Antibiotic overuse: Stop the killing of beneficial bacteria. Nature 476:393-4
Bragina A, Berg C, Müller H, Moser D, Berg G (2013) Insights into functional bacterial diversity and its effects on Alpine bog ecosystem functioning. Sci Rep. 3:1955
Caporaso JG, Lauber CL, Walters WA, Berg-Lyons D, Huntley J, Fierer N, et al (2012) Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. ISME J 6:1621-4
Caputo F, Nicoletti F, Picione FDL, Manici LM (2015) Rhizospheric changes of fungal and bacterial communities in relation to soil health of multi-generation apple orchards. Biological Control 88:8-17
Carisse O (2000) 50 years of research on biological control. Integrated control of pome fruit diseases. IOCB WPRS Bull 23:5-10
Erlacher A, Cardinale M, Grosch R, Grube M, Berg G (2014) The impact of the pathogen Rhizoctonia solani and its beneficial counterpart Bacillus amyloliquefaciens on the indigenous lettuce microbiome. Frontiers in microbiology, 5
Fiss M, Kucheryava N, Schönherr J, Kollar A, Arnold G, Auling G (2000) Isolation and characterization of epiphytic fungi from the phyllosphere of apple as potential biocontrol agents against apple scab (Venturis inaequalis). Zeitschrift für Pflanzenkrankheiten und Pflanzenschutz: 1-11
Jedrychowski W, Maugeri U (2009) An apple a day may hold colorectal cancer at bay: recent evidence from a case-control study. Rev Environ Health 24, 59-74
Köhl J, Scheer C, Holb IJ, Masny S, Molhoek W (2015) Toward an integrated use of biological control by Cladosporium cladosporioides H39 in apple scab (Venturia inaequalis) management. Plant Disease, 99(4), 535-543
Le Chatelier E, Nielsen T, Qin J, et al. (2013) Richness of human gut microbiome correlates with metabolic markers. Nature 29: 541–546
Leff JW, Fierer N (2013) Bacterial communities associated with the surfaces of fresh fruits and vegetables. PLoS One. 8: e59310
Lozupone CA, Stombaugh JI, Gordon JI, Jansson JK, Knight R (2012) Diversity, stability and resilience of the human gut microbiota. Nature 489:220-30
MacHardy WE (1996) Apple scab: biology, epidemiology, and management. American Phytopathological Society (APS Press)
Muyzer G, Smalla K. 1998. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology. Antonie Van Leeuwenhoek73:127-41
Perez A, Lin BH, Allshouse J (2001) Demographic Profile of Apple Consumption in the United States. In Fruit and Tree Nuts SO, pp 37-47
Philippot L, Raaijmakers JM, Lemanceau P, van der Putten WH. 2013. Going back to the roots: the microbial ecology of the rhizosphere. Nat Rev Microbiol 11:789-99
Qin J, Li R, Raes J, Arumugam M, Burgdorf K, Solvsten M. et al. (2010) A human gut microbial gene catalogue established by metagenomic sequencing. Nature 464: 59–65
Rybakova D, Schmuck M, Wetzlinger U, Varo Suarez A, Murgu O, Müller H, Berg G (2015b) Kill or cure? The interaction between endophytic Paenibacillus and Serratia strains and the host plant is shaped by plant growth conditions. Plant and soil:1-15
Shade A, McManus PS, Handelsman J (2013) Unexpected diversity during community succession in the apple flower microbiome. mBio 4 (2): e00602-12
Thomas F, Hehemann JH, Rebuffet E, Czjzek M, Michel G (2011) Environmental and gut bacteroidetes: the food connection. Front Microbiol. 30: 2-93
Weiss A, Mögel G, Kunz S (2006) Development of“ Boni-Protect“-a yeast preparation for use in the control of postharvest diseases of apples. In Ecofruit-12th International Conference on Cultivation Technique and Phytopathological Problems in Organic Fruit-Growing: Proceedings to the Conference from 31st January to 2nd February 2006 at Weinsberg/Germany (pp. 113-117). Fördergemeinschaft Ökologischer Obstbau eV (FÖKO)
Yepes LM, Aldwinckle HS (1993) Selection of resistance to Venturia inaequalis using detached leaves from in vitro-grown apple shoots. Plant Science 93(1-2):211-216